北化新闻网10月31日电 近日,吴边/崔颖璐团队在《科学》(Science)杂志发表题为”Glycolysis-compatible urethanases for polyurethane recycling“的研究论文。研究团队借助人工智能驱动的微生物酶资源挖掘策略,成功发现了一种新型脲酶AbPURase。该酶具有优异的有机溶剂耐受性,在高浓度醇解反应介质中仍能保持极高催化活性与稳定性,与工业聚氨酯醇解工艺高度兼容。这一成果首次实现了在工业条件下聚氨酯的规模化生物解聚,为聚氨酯塑料的绿色循环利用提供了高效、可持续的新路径。
本项目由北京化工大学与中科院微生物所联合完成,吴边教授和崔颖璐研究员为论文共同通讯作者,特别研究助理陈艳春、博士研究生孙瑨原和石珂伦为共同第一作者。本研究获得了国家重点研发计划、国家杰出青年科学基金和国家优秀青年科学基金等项目的资助。
聚氨酯(PU)塑料作为全球第六大常用聚合物,年产量超过3000万吨,凭借其优异的物理化学性能,已广泛应用于国民经济的多个领域。其产品涵盖建筑隔热与保温材料,生活用品(如床垫、沙发垫材,冰箱与空调的隔热层等),以及交通运输工具(如汽车、飞机座椅等部件)等诸多领域。然而,随着其应用范围和产能规模的持续扩大,聚氨酯废弃物对环境造成的压力日益加剧,推动废旧聚氨酯塑料的资源化再生技术开发已成为当务之急。作为一种高分子聚合物,聚氨酯材料主要由异氰酸酯与聚醚多元醇反应制得。理论上,可通过水解等化学方法将其解聚,回收原始单体实现循环利用。然而,由于聚氨酯具有高度交联的三维网络结构,导致其解聚过程条件苛刻,且难以实现单体的高效、完全回收,严重制约了其闭环再生的产业化进程。
为实现聚氨酯的生物酶解,关键在于开发能够高效水解氨基甲酸酯键的脲酶。然而,现有的EC酶分类系统中,对这类脲酶的分类标注存在缺失。目前已知的少数可水解芳香类氨基甲酸酯的脲酶,在传统生物信息学分类中均被标识为脂肪酶或酰胺酶。因此,研究团队基于前期自主开发的蛋白质Pythia预训练模型(The Innovation 2025, 100750, https://pythia.wulab.xyz),结合监督学习与自监督学习,进一步开发了基于图神经网络的酶挖掘工具GRASE(GNN-based Recommendation of Active and Stable Enzymes)。该工具通过估计蛋白质序列与结构之间的似然性,预测蛋白质稳定性;并利用图神经网络提取催化口袋中氨基酸的局部微环境与整体结构信息,通过在高维空间中计算不同催化口袋特征向量之间的余弦相似度以度量其功能相似性,从而实现对酶底物特异性与反应偏好性的精细推断。
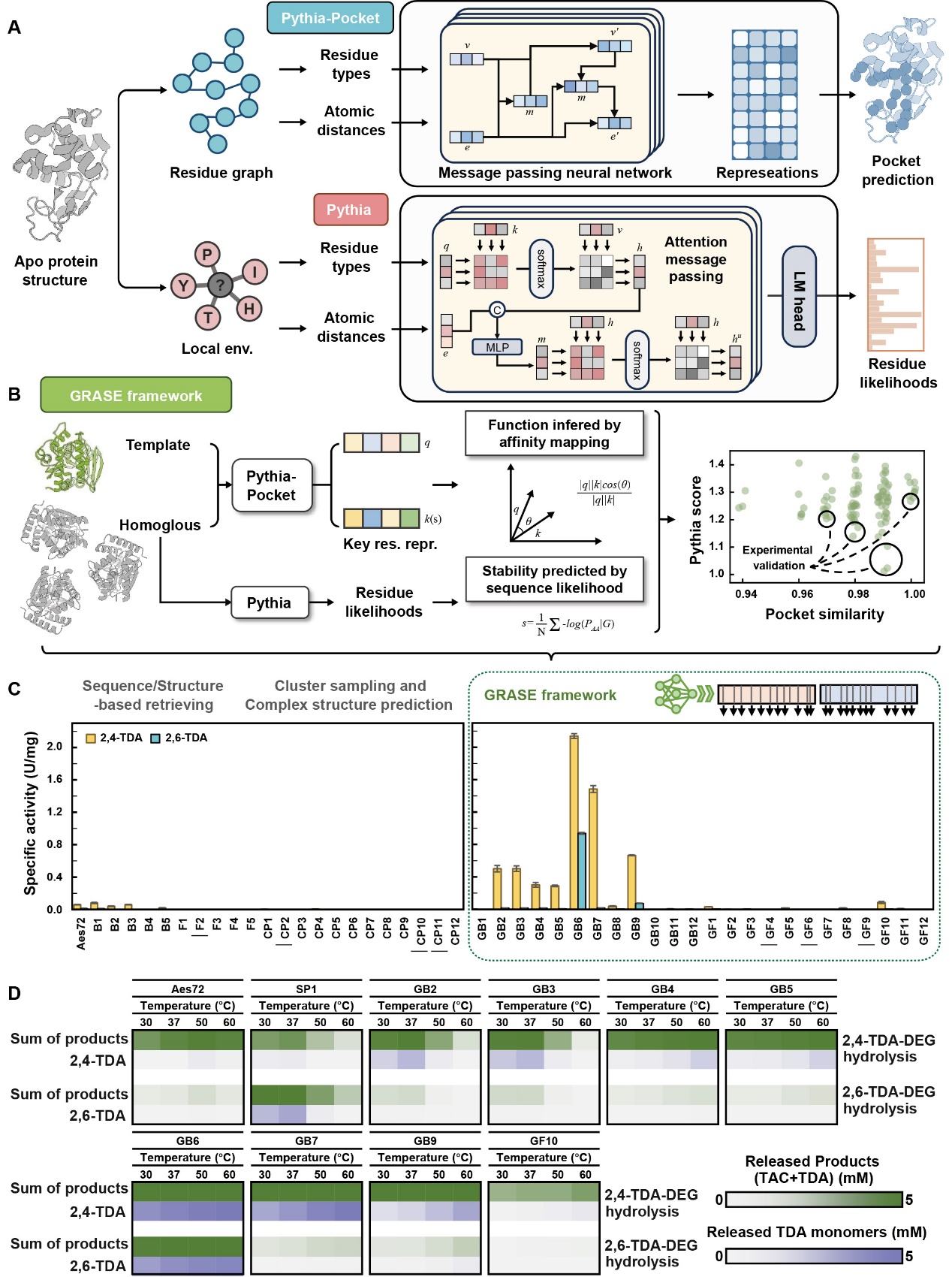
图1:GRASE模型架构图
团队利用GRASE工具,从宏基因组数据中成功挖掘到多个具有氨基甲酸酯降解活性的酶,其中来源于脂环酸芽孢杆菌属(Alicyclobacillus sp.)的AbPURase酶表现出全方位的优异特性。该酶具有一个V形活性口袋,该结构由一个紧密的疏水核心及多个脯氨酸残基所稳定的盖结构域构成。这一独特的结构不仅有助于酶与底物的结合,还有效阻隔了有机溶剂分子向活性中心的渗透,从而保障了AbPURase在高浓度有机溶剂中结构和功能的完整性。
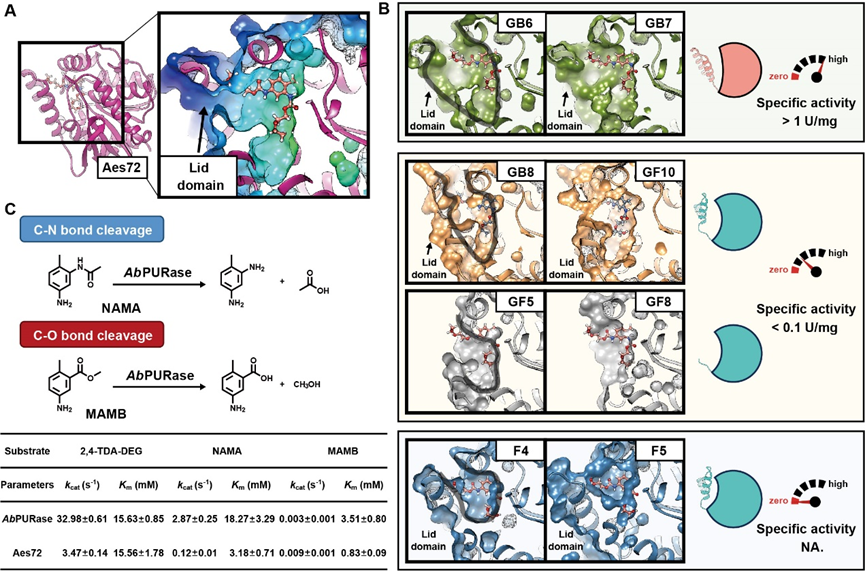
图2:新开发脲酶的结构解析
得益于其独特的天然结构特性,AbPURase酶在工业醇解工艺中的高浓度二乙二醇环境中,活性高达已报道脲酶的465倍,显示出卓越的催化效率和极强的有机溶剂耐受性。在模拟工业化反应条件(底物载量500 g/L)的千克级商业聚醚型聚氨酯泡沫降解实验中,AbPURase在8小时内实现了98.6%的底物转化率,降解单体TDA及醇解剂DEG的回收率分别高达94.7%和98.5%。此外,AbPURase还表现出优异的操作稳定性,可在多轮循环中维持高降解活性,显示出该酶的工业应用潜力。
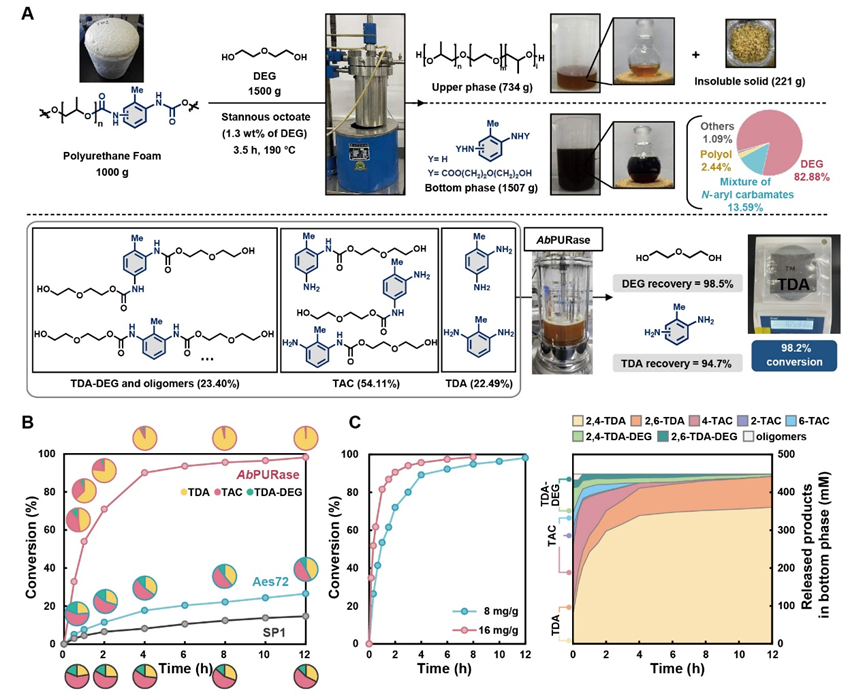
图3:商业级聚氨酯的酶法醇解
总体而言,目前人类已知的上亿条蛋白质序列中蕴藏着丰富的生物学规律与演化信息。然而传统酶挖掘方法通常依赖序列相似性或粗粒化的酶学分类号进行筛选,导致大量序列相似度低但功能相似的酶资源被系统性遗漏。研究团队开发的GRASE系统有效突破了传统序列分析方法的局限,通过深度学习捕捉蛋白质局部结构的高维特征,高效挖掘自然界中隐藏的微生物酶资源“暗物质”。该研究不仅为塑料循环利用提供了具备工业应用潜力的解决方案,更推动了人工智能在工业酶开发领域的智能化进程。
论文链接:https://www.science.org/doi/10.1126/science.adw4487
吴边教授简介
吴边,北京化工大学生命科学与技术学院教授,国家杰出青年基金获得者(2023),国家重点研发计划项目负责人、指南专家组成员,中国微生物学会酶工程专委会主任。主要从事生物催化研究。近年来,聚焦酶元件挖掘、机制解析与工程改造,将人工智能技术应用于酶工程领域,推动了生物大分子设计的发展。在Science, Nature Catalysis, Nature Chemical Biology等学术刊物上发表论文。研发的多肽药物酶法拼装、β-氨基酸生物合成、氮杂环类药物中间体顺次发酵等多项技术成功实现产业化应用。获河北省科技进步一等奖、中和青年多肽科学家奖与中国微生物学会优秀青年酶工程学家奖。
责编:王雨晴 刘一君 梁燕亮